Machine Learning - 7 astuces pour scaler Python sur de grands datasets
Python est le langage privilégié chez les Data Scientists, notamment grâce à toutes ses librairies open-source et sa facilité de mise en production du code. Pourtant, à mesure que la volumétrie des données augmente, le passage à des paradigmes différents comme ceux de Spark et Hadoop est recommandé car plus scalable. Cependant, cela nécessite souvent de mettre en place une infrastructure et d’adapter son code. Voici quelques astuces qui permettent d’étendre l’utilité de Python pour des datasets de plusieurs gigaoctets dans un contexte mono-machine.
1 - Supprimer les variables inutiles
Les premières optimisations que l’on peut faire sont au niveau de la mémoire. De fait, on consomme souvent plus de RAM que ce dont on a besoin.
Par exemple, les variables non utilisées peuvent être supprimées au fur et à mesure. Pour cela, on peut utiliser del.
Note : pour la suite, df correspond à un DataFrame sur Pandas.
del df
Une commande HTOP sur un terminal permet de vérifier que l’espace a bien été libéré.
2 - Supprimer les colonnes inutiles
De même, les colonnes inutiles peuvent être supprimées au fur et à mesure.
La première façon de le faire est de sélectionner soi-même ses colonnes, en créant une liste :
features_cols = ['col1', 'col2', 'col3']
df = df[features_cols]
Outre faire soi-même sa sélection de colonnes, on peut aussi utiliser des astuces pour supprimer certaines colonnes automatiquement en amont ; par exemple, celles qui ne contiennent que des NAs :
df = df.dropna(axis=1, how='all')
Idem, avec les colonnes qui ne contiennent qu’une valeur unique :
df = df.loc[:, (df != df.iloc[0]).any()]
Nous pouvons supprimer les colonnes qui ne contiennent, au contraire, que des valeurs distinctes :
for col in df.columns:
if len(df[col].unique()) == len(df):
df = df.drop(col, axis=1)
Cette technique est à prendre avec des pincettes pour deux raisons. La première est que l’on risque de supprimer les colonnes numériques ayant des précisions si grandes que toutes les valeurs sont distinctes. La deuxième est que ces colonnes n’ayant que des valeurs distinctes sont parfois sensées, comme par exemple les dates qui peuvent porter un signal temporel.
Nous pouvons aussi supprimer les colonnes dupliquées, c’est-à-dire qui contiennent exactement les mêmes valeurs :
_, duplicated_cols_idx = np.unique(df, axis=1, return_index=True)
df = df.iloc[:, duplicated_cols_idx]
Enfin, nous pouvons supprimer les colonnes dupliquées à une transformation près : par exemple, si la colonne 1 a pour valeurs (“a”, “b”, “a”, “c”) et la colonne 2 a pour valeurs (“category_1”, “category_2”, “category_1”, “category_3”), les colonnes sont dupliquées puisque “a” “équivaut à “category_1”, “b” équivaut à “category_2” et “c” équivaut à “category_3”.
Pour s’affranchir de ces colonnes, une façon de faire est la suivante :
df["column_1"] = df["column_1"].astype("category").cat.codes
df["column_2"] = df["column_2"].astype("category").cat.codes
_, duplicated_cols_idx = np.unique(df, axis=1, return_index=True)
df = df.iloc[:, duplicated_cols_idx]
Les colonnes “column_1” et “column_2” sont transformées en type category puis en chiffres grâce à .cat.codes . Cette transformation permet de transformer les valeurs rencontrées en chiffre, de façon séquentielle. Ainsi, si nous reprenons l’exemple précédent, la colonne 1 deviendra (0, 1, 0, 2) puisque “a” est transformé en 1 car rencontré en premier, “b” est transformé en 2 car rencontré en deuxième, “c” est transformé en 3 car rencontré en troisième.
Or, cette transformation va aussi transformer la colonne 2 en (0, 1, 0, 2). Dès lors, nous pouvons appliquer les deux lignes permettant de supprimer les colonnes dupliquées.
3 - Supprimer les lignes inutiles
Une autre optimisation est de supprimer les lignes inutiles. J’ai déjà rencontré un cas où un out of memory était provoqué lors d’une jointure (merge) entre deux DataFrames ; cela est dû au fait que lors de la jointure, les deux DataFrames de gauche et de droite et le DataFrame résultat étaient chargés en mémoire !
Après cela, je me suis rendu compte que certaines lignes n’étaient pas joignables : en effet, certaines clés n’étant pas communes aux deux DataFrames, lors d’un inner merge , les lignes correspondant à ces clés disparaissent automatiquement. Or, comme expliqué précédemment, Python alloue la mémoire des deux DataFrames à joindre et du DataFrame résultat. Autrement dit, supprimer les lignes en amont de la jointure - qui seront de toute façon supprimées lors de la jointure - fera gagner de l’espace mémoire lors de cette étape.
Sur la figure 1, une jointure de df1 et df2 est réalisée respectivement sur df1_key et df2_key. Nous remarquons qu’en effectuant cette opération, les valeurs 3 de df1_key et 4 de df2_key sont supprimées.
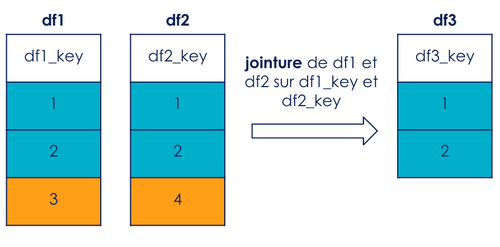
Nous pouvons donc supprimer ces valeurs en amont de la jointure.
Voici un exemple de code permettant de mettre en application cela. Si je veux joindre mes DataFrames df1 et df2 respectivement sur les clés df1_key et df2_key, je sélectionne d’abord les valeurs des clés communes (intersection_keys) puis filtre mes deux DataFrames grâce à ces clés communes :
intersection_keys = set(df1[df1_key]).intersection(set(df2[df2_key]))
df1 = df1[df1[df1_key].isin(intersection_keys)]
df2 = df2[df2[df2_key].isin(intersection_keys)]
Que l’on soit bien clair : le résultat de la jointure sera le même ; seulement, supprimer ces lignes en amont permet d’économiser de la RAM au moment propice. Il est possible d’effectuer le même traitement si la jointure est effectuée sur plusieurs colonnes.
4 - Downcaster les variables
Les variables, en particulier les Series des DataFrames, utilisent souvent plus de mémoire que nécessaire. Or, un gain vraiment significatif peut être effectué à ce niveau : comme expliqué dans l’article “Using Pandas with large data” de Dataquest, les Series de type Object (donc des strings) prennent bien plus d’espace mémoire que les Series de type Category, type introduit dans Pandas 0.15. De même, pourquoi allouer 64 bits pour des nombres qui vont de 1 à 100 lorsque nous pourrions utiliser 8 bits ?
Il y a plusieurs raisons pour lesquelles les types peuvent ne pas être les bons :
- Les types inférés par Pandas lors de la lecture d’un fichier ne sont pas forcément les bons (ex : Pandas peut choisir un type ‘object’ au lieu d’un type ‘category’, un int16 à la place d’un int8, etc.)
- Il arrive que certaines opérations transforment le type des variables
- Lorsqu’une nouvelle Series est créée, Pandas prend souvent un type qui consomme plus que nécessaire (ex : si je crée une Series composée des valeurs 1, 2, 3, le type de ma Series sera int64, alors qu’un uint8 ou int8 eût été largement suffisant !)
Il est possible de “forcer” le casting en utilisant .astype(), par exemple en int8 :
df['column_1'] = df['column_1'].astype('int8')
Sinon, voici un exemple de code permettant de downcaster les variables numériques de façon automagique. Les colonnes de type int et float sont sélectionnées, puis downcastées respectivement au type int ou float consommant le moins de mémoire possible :
int_columns = df.select_dtypes(include=['int']).columns.tolist()
float_columns = df.select_dtypes(include=['float']).columns.tolist()
df[int_columns] = df[int_columns].apply(pd.to_numeric, downcast='integer')
df[float_columns] = df[float_columns].apply(pd.to_numeric, downcast='float')
A noter que si vous êtes sûr d’avoir des valeurs positives, utilisez des entiers non signés (uint) plutôt que des entiers (int) : par exemple, les uint8 vont de 0 à 255 tandis que les int8 vont de -128 à 127. La documentation de NumPy permet d’avoir une vue exhaustive sur les types de données numériques.
En ce qui concerne les Series de type Object, le gain est stupéfiant lorsqu’elles sont castées en Category. Essayez donc de vous en affranchir le plus vite possible, soit en les supprimant lorsque vous n’en avez plus besoin, soit en les castant en Category s’il s’agit de variables catégorielles. Par exemple :
df['column_1'] = df['column_1'].astype('category')
Le gain est surtout important pour les colonnes à faibles cardinalités. Il ne faut pas oublier non plus que la moindre transformation post-casting, par exemple en exécutant la commande suivante, fait re-basculer la Serie en object :
df['column_1'] = df['column_1'].str.lower()
5 - Stocker ses fichiers en Parquet
Parquet est un format orienté colonne utilisé par l'écosystème Apache Hadoop, notamment Spark. Le format Parquet est également disponible sur Pandas depuis la version 0.21. Non seulement l’espace sur disque est moindre, mais les I/O sont bien plus rapides.
Si vous vous peinez à lire un gros fichier csv, le meilleur conseil que je peux vous prodiguer est :
- Lisez votre csv (long)
- Stockez le en Parquet
Aussi simple que cela. Stocker un fichier une bonne fois pour toutes en Parquet, ça envoie du bois (pun intended) : il sera bien plus rapide à la lecture, et c’est de toute façon transparent pour vous.
df = pd.read_csv(raw_data_path+file_path)
df.to_parquet('{}/data_parquet.gzip'.format(interim_data_path))
df = pd.read_parquet('{}/data_parquet.gzip'.format(interim_data_path))
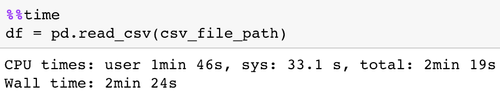
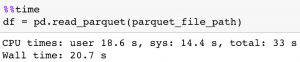
Pour s’en convaincre, voici l’exemple d’une lecture d’un fichier .csv qui prend 4.4 go sur disque. Le fichier .csv prend plus de 2 min à être lu :
Une fois stocké en Parquet, le fichier ne prend plus que 928 mo sur disque. En plus de cela, sa lecture n’est plus que de 20 secondes !
6 - Utiliser Dask plutôt que Pandas
Dask permet de paralléliser les calculs en proposant une API similaire à 95% à Pandas. L’avantage principal est de pouvoir passer de DataFrames Pandas à des DataFrames Dask sans avoir à changer grand chose. Par exemple :
import dask.dataframe as dd
df_dask = dd.read_parquet('{}/data_parquet.gzip'.format(interim_data_path))
df_dask_column_1_max = df_dask.groupby(['column_1']).max()
Comme nous pouvons le remarquer, les fonctions appelées ci-dessus sont similaires en tous points à ce que nous ferions en Pandas.
Dask a beaucoup d’atouts, et est en particulier rapide pour calculer des jointures, des agrégats, des filtres, etc.
Nonobstant cela, le fait que Dask ait un comportement lazy déporte certaines lenteurs de Pandas à d’autres endroits, ce qui n’est pas toujours souhaitable.
Je m’explique : lire un fichier Parquet en Dask sera immédiat (car les données ne seront pas réellement chargées en mémoire à ce moment), tandis que lire un fichier Parquet en Pandas prendra quelques secondes à quelques minutes selon la taille de mon fichier. En revanche, en phase d’exploration, j’ai souvent besoin d’utiliser des .head(), des .shape ou autres informations sur un DataFrame. En l’occurrence, ces opérations sont bien plus longues à effectuer sur Dask, puisque les données sont réellement chargées en mémoire à ce moment seulement. Par exemple, en affichant les 5 premières lignes d’un DataFrame, par l’intermédiaire de Pandas et de Dask, toutes choses étant égales par ailleurs :
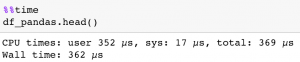
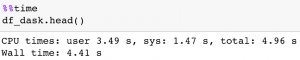
De fait, ces opérations sont bien plus rapides en Pandas, et à mesure qu’elles sont répétées en phase d’exploration, le gain est important.
Similairement, effectuer un agrégat sur Pandas peut prendre du temps :
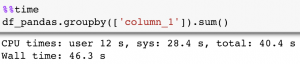
Alors que sur Dask, effectuer un agrégat est immédiat , parce que son évaluation est lazy :

Mais pour réellement effectuer le calcul et pouvoir afficher le résultat, il faut appeler la méthode .compute(). Cette fois l’opération est plus chronophage sur Dask que sur Pandas:
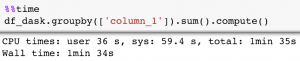
Utiliser Dask peut donc être un peu déroutant au début, mais des gains substantiels peuvent être obtenus lorsque plusieurs opérations arithmétiques, jointures, agrégats, etc. sont chaînés et en n’utilisant un .compute() qu’à la fin.
Finalement, j’utiliserais plutôt Pandas lorsque mon DataFrame peut tenir en mémoire facilement ou si je suis en phase exploratoire. Une fois qu’un fichier est lu une bonne fois pour toutes, cela nous permet d’obtenir des résultats interactifs, c’est confortable.
Dask a plutôt un pied dans la zone grise, entre le small data (Pandas) et le huge data (Spark & co), c’est-à-dire pour le cas où l’on a besoin d'une meilleure gestion des ressources locales quand la donnée n'est pas encore trop importante au point d'être stockée en distribué.
Dans la documentation d'introduction de Dask il est fait référence à une exécution en cluster, qui permettrait d'aller un cran plus loin encore, voire d'empiéter sur le territoire des Spark & co en tout cas pour la partie Data Processing.
7 - Choisir un modèle adapté
Certains modèles de Machine Learning scalent mieux que d’autres. En ce qui concerne les implémentations d’algorithmes de Boosting, XGBoost, que j’utilise très régulièrement, se fait détrôner par LightGBM à mesure que les données grandissent, pour des performances semblables en terme de précision.
Il suffit de s’en convaincre en regardant le graphe en figure 1, sur lequel nous observons sur une échelle log-log le temps pris par les deux algorithmes pour converger, en fonction du nombre de lignes. Tous les paramètres sont égaux par ailleurs, à savoir :
- la machine et les ressources utilisées
- les données en entrée (qui sont des données prises au hasard : en prenant d’autres données, les résultats pourraient légèrement varier)
- les paramètres des deux algorithmes
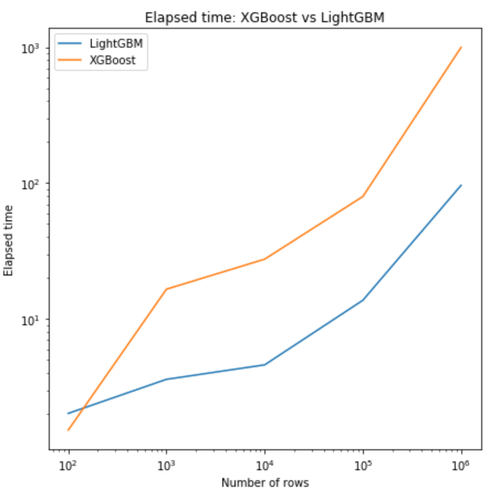
Si LightGBM est à peine en deçà de XGBoost sur 100 lignes, il est 5 fois plus rapide sur 1000 lignes et 10 fois plus rapide sur 100 000 lignes !
En outre, les paramètres sont similaires sur les deux APIs, il suffit juste d’adapter les noms des paramètres. Par exemple, sur XGBoost, un exemple de paramètres pourrait être :
xgb_params = {
'objective': 'multi:softmax',
'eval_metric': 'mlogloss',
'num_class': 100,
'eta': 0.15,
'max_depth': 10,
'subsample': 0.6,
'colsample_bytree': 0.7,
'seed': seed,
'n_jobs': -1
}
L'équivalent sur LightGBM serait :
lgb_params = {
'boosting_type' : 'gbdt',
'objective' :'multiclass',
'metric': 'multi_logloss',
'num_class': 100,
'learning_rate' : 0.15,
'num_leaves' : 10,
'bagging_fraction': 0.6,
'feature_fraction': 0.7,
'bagging_freq':1,
'seed': seed,
'n_jobs': -1
}
Donc, lorsque la volumétrie augmente, autant privilégier LightGBM à XGBoost.
A noter qu'une troisième implémentation performante du boosting, Catboost développé par Yandex, peut être utilisée. Certains benchmarks montrent que dans certains cas, Catboost peut être encore plus rapide que LightGBM.
En plus de cela, il faut toujours s’assurer que la complexité du modèle est nécessaire : pour le cas des modèles à base d’arbres par exemple, est-ce que le modèle pourrait performer autant avec une profondeur plus faible ou un nombres d’arbres réduit ?
Conclusion
A mesure que la volumétrie devient importante, l’idée d’un passage à l’écosystème Hadoop et Spark commence à germer. Mais force est de constater que changer d’écosystème veut dire changer d’infrastructure, de code, de paradigme, et ces risques peuvent facilement nous faire tomber de Charybde en Scylla.
A ces égards, il est souvent possible d’utiliser des machines comportant plus de RAM, plus de CPU/GPU, plus de stockage et d’optimiser son code Python. Ces 7 astuces non exhaustives, utilisées ensemble ou non, permettent de rester sur Python, Pandas & co un peu plus longtemps.